中国团队发现新蝙蝠冠状病毒,力证新冠起源于自然
摘要:新的一项研究显示,去年在云南勐腊县获得的蝙蝠样本中发现一种新冠状病毒,该病毒在最长编码基因区1ab与新冠病毒(SARS-CoV-2)一致性达到97.2%。同时,研究团队在这一新冠状病毒中S蛋白(刺突糖蛋白)S1和S2交界处也发现插入了三个残基PAA,他们由此认为SARS-CoV-2在S1和S2的交界处的四氨基酸插入并非“人工痕迹”,有力证明了其起源于自然。
最新的一项研究显示,去年在云南勐腊县获得的蝙蝠样本中发现一种新冠状病毒,该病毒在最长编码基因区1ab与新冠病毒(SARS-CoV-2)一致性达到97.2%。同时,研究团队在这一新冠状病毒中S蛋白(刺突糖蛋白)S1和S2交界处也发现插入了三个残基PAA,他们由此认为SARS-CoV-2在S1和S2的交界处的四氨基酸插入并非“人工痕迹”,有力证明了其起源于自然。
当地时间5月10日,《细胞》(Cell)旗下子刊《现代生物学》(Current Biology)在线发表了“A novel bat coronavirus closely related to SARS-CoV-2 contains natural insertions at the S1/S2 cleavage site of the spike protein ”,来自山东第一医科大学、山东省高等学校新发传染病病因流行病学实验室、中国科学院西双版纳热带植物园、中国科学院北京生命科学研究所、中国科学院武汉病毒研究所、中国科学院微生物研究所、澳大利亚悉尼大学联合团队的研究人员给出了上述结论。

这篇论文中,研究团队报告了一种新的蝙蝠来源冠状病毒,命名为RmYN02,是在2019年5月至10月期间从中国云南省收集的227只蝙蝠的基因组分析中鉴定出来的。
研究显示,RmYN02和新型冠状病毒SARS-CoV-2在全病毒基因组中的同源性为93.3%,在与SARS-CoV-2最接近的1ab基因中的同源性为97.2%。相比之下,RmYN02受体结合域(RBD)与SARS-CoV-2之间呈现低序列同源性(61.3%),可能不与血管紧张素转换酶2(ACE2)结合。
然而,与SARS-CoV-2相似,RmYN02的特点是在S蛋白的S1和S2亚单位的剪切位点处有多个氨基酸插入。研究团队认为,这就有力证明了这种插入事件在自然界中是可以发生的。
这些数据表明,SARS-CoV-2源于蝙蝠和其他野生动物中存在的病毒之间的多重自然重组。
该论文通讯作者为山东第一医科大学基础医学院病原生物学研究所所长、山东省高等学校新发传染病病因流行病学实验室主任史卫峰,中国科学院微生物研究所病毒传播预警与致病机制研究组组长、项目研究员毕玉海,中国科学院西双版纳热带植物园综合保护中心景观生态组组长、副教授Alice Catherine Hughes。该研究此前于当地时间3月5日发表在预印本网站bioRxiv上。
新冠病毒起源尚不明确,野生动物中仍有大量冠状病毒
SARS-CoV-2在中国及其他地区造成了前所未有的肺炎流行,已引起全球范围的公共卫生关注。尽管蝙蝠被认为是SARS-CoV-2 最有可能的自然宿主,但病毒的起源仍不清楚。
系统发育分析表明,SARS-CoV-2是一种不同于SARS-CoV和MERS-CoV的新型冠状病毒。到目前为止,与SARS-CoV-2最密切相关的病毒是RaTG13,它是中科院武汉病毒所石正丽团队于2013年在云南采集的一只中华菊头蝠标本中分离发现。RaTG13病毒株和新冠病毒具有96.1%的核苷酸同源性、92.9%的S基因同源性。这些数据再次说明蝙蝠是冠状病毒的重要宿主。
然而,值得注意的是,香港大学公共卫生学院新发传染病国家重点实验室管轶教授、广西医科大学胡艳玲教授团队,以及华南农业大学、岭南现代农业科学与技术广东省实验室沈永义教授、肖立华教授团队两个研究小组此前均报告了存在于马来穿山甲的SARS-CoV-2相关冠状病毒,这些穿山甲通过非法走私进入广西和广东。
研究团队提到,尽管在全基因组水平上,这些穿山甲中检测到的冠状病毒与SARS-CoV-2的距离要远于RaTG13与SARS-CoV-2的距离,但它们在S蛋白的受体结合域(RBD)上与SARS-CoV-2非常相似。
因此,尽管目前还不清楚穿山甲是否是SARS-CoV-2传播到人过程中的中间宿主,但它们可能在冠状病毒的生态学和进化中发挥重要作用。
他们认为,这些在穿山甲中发现的病毒可以表明,在野生动物中仍有大量的冠状病毒样本,其中一些可能直接参与了SARS-CoV-2的出现。

SARS-CoV-2和几种代表性蝙蝠来源冠状病毒序列比较。
一种新的蝙蝠来源冠状病毒,命名RmYN02
论文中提到,2019年5月至10月,研究团队从云南勐腊县的227只蝙蝠中一共收集了302个样本。这些蝙蝠属于20个不同种类,大多数样本从马来菊头蝠Rhinolophus malayanus(n=48, 21.1%)、中蹄蝠Hipposideros larvatus (n=41,18.1%)和小褐菊头蝠Rhinolophus stheno(n=39,17.2%)中获得。样本来源于多种组织,包括翼膜(219)、肺(2)、肝(3)和粪便(78)。
除了3只蝙蝠外,其余所有蝙蝠均为在活着时取样并被释放。
利用新一代宏基因组测序技术,研究团队首先锁定了2个初步的一致序列。这些序列产生的样本来自于2019年5月6日至7月30日期间的11份马来菊头蝠粪便。经过一系列验证步骤,研究团队得到一个部分(23395bp)和一个完整(29671bp)的蝙蝠冠状病毒基因组序列,分别命名为BetaCoV/Rm/Yunnan/YN01/2019 (RmYN01)和BetaCoV/Rm/Yunnan/YN02/2019 (RmYN02)。
相比之下,RmYN02与SARS-CoV-2密切相关,表现出93.3%的核苷酸序列一致性,但从全长基因组层面来说,RaTG13和SARS-CoV-2的一致性更高(96.1%)。RmYN02和SARS-CoV-2在大多数基因组区域(如1ab、3a、E、6、7a、N和10)非常相似(>96%序列一致性)。特别是,RmYN02在最长编码基因区1ab (n=21285)与SARS-CoV-2一致性达到97.2%。
不过,RmYN02和SARS-CoV-2在S基因中的序列一致性(核苷酸71.8%,氨基酸72.9%)远低于RaTG13和SARS-CoV-2之间的97.4%。另外值得注意的是,RmYN02和SARS-CoV-2在RBD中的氨基酸同源性仅为62.4%。而来自广东的穿山甲冠状病毒和SARS-CoV-2在RBD中的氨基酸同源性为97.4%,也是在RBD区域目前和SARS-CoV-2最接近的。
围绕SARS-CoV-2的同源模型、体外实验和S蛋白的三维结构分析结果都表明,SARS-CoV-2和SARS-CoV一样,也可以利用ACE2作为细胞受体。研究团队也同样使用同源模型分析了RmYN02、RaTG13和两个穿山甲CoVs的RBD。
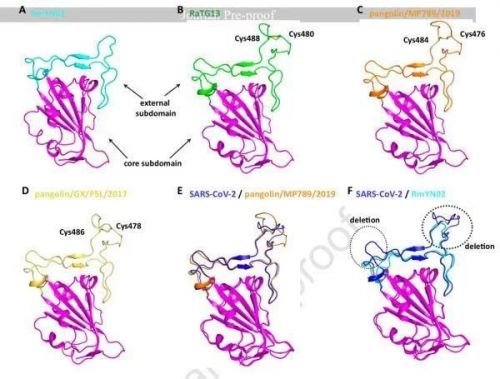
RmYN02和代表性冠状病毒RBD结构的同源建模和结构比较。
研究发现,RmYN02 RBD中的氨基酸缺失在受体结合位点附近形成了两个比SARS-CoV-2 RBD短的环。重要的是,在SARS-CoV、SARS-CoV-2、RaTG13、穿山甲/MP789/2019、穿山甲/GX/P5L/2017的外子域(external subdomain )保守的二硫键在RmYN02中缺失了。研究团队推测,这些缺失可能导致构象变化,从而减少RmYN02 RBD与ACE2的结合,甚至导致不结合。
当然也有可能的一种情况是,包括RmYN02、ZXC21和ZC45在内的环缺失的SARS相关冠状病毒,使用了一种我们目前未知的受体。
值得一提的是,此前有研究认为,RBD中的6个氨基酸残基(L455、F486、Q493、S494、N501和Y505)是SARS-CoV-2与ACE2有效受体结合的主要决定因素。与同源建模一致,穿山甲/MP789/2019在所有6个位置上都具有与SARS-CoV-2相同的氨基酸残基。相比之下,RaTG13、RmYN02和RmYN01与SARS-CoV-2均只在1个位置上有相同的氨基酸残基。
研究团队认为,这种进化模式是重组和自然选择的复杂结合的表现。
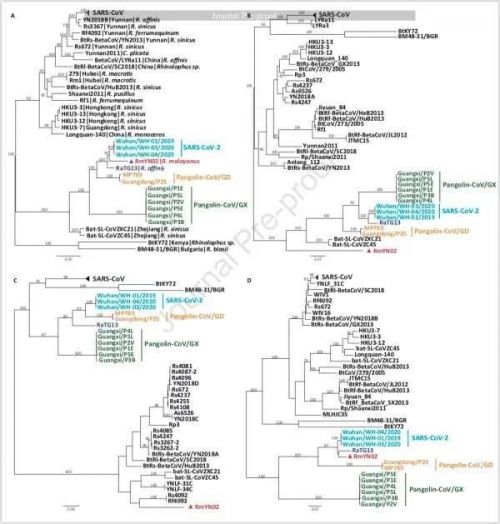
系统发育树:A 全长基因组;B、S基因;C、RBD ;D、RdRp(RNA依赖的RNA聚合酶
研究团队还对RmYN02、RaTG13、SARS-CoV-2和穿山甲中的蝙蝠冠状病毒进行了系统发育分析。与先前的研究相符,穿山甲beta-CoVs形成两个亚型。然而,论文中提到,穿山甲是否是这些病毒的天然蓄水池,或者它们从蝙蝠或其他野生动物中独立获得,这都需要进一步验证。
更值得注意的是,在大多数病毒基因组中,RmYN02与SARS-CoV-2的亲缘关系最近,尽管这两种病毒之间仍然有一段较长的分支距离。S基因树显示,SARS-CoV-2离RaTG13较近,和RmYN02较远,这表明后者在S基因经历了重组。在RBD的系统发育树上,SARS-CoV-2和pangolin-CoV/GD最密切相关,与蝙蝠病毒都较远,再次表明重组发生。最后,完整的RNA依赖的RNA聚合酶(RdRp)基因(在RNA病毒的系统发育分析中经常被使用)系统发育分析显示,RmYN02、RaTG13和SARS-CoV-2形成了一个与穿山甲病毒完全不同的亚群。
SARS-CoV-2是自然起源,可能通过重组获得
类似禽流感病毒(AIVs)血凝素(HA)蛋白的方式,冠状病毒的S蛋白在功能上分裂成两个亚基S1和S2。而在某些AIV亚型的剪切位点上插入多碱基氨基酸被认为与增强致病性有关。
值得注意的是,SARS-CoV-2的特征之一即为在S1和S2的交界处有一个四氨基酸的插入,这在其他β冠状病毒的其他谱系中没有观察到过。这种被称为弗林蛋白酶剪切位点的插入是SARS-CoV-2独有的,目前在所有检测的SARS-CoV-2序列中都发现存在。
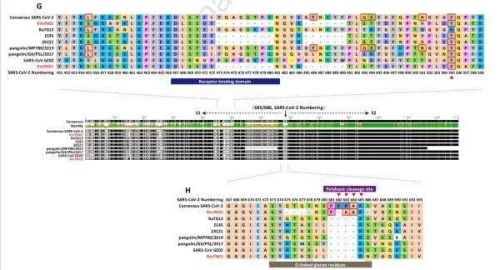
研究团队此番在RmYN02中S1和S2交界处也发现插入了三个残基PAA,他们认为这是非常重要的。“虽然SARS-CoV-2插入的残基(以及由此产生的核苷酸)与RmYN02中的残基不同,但可以表明它们是一个独立的插入事件,它们在野生动物(蝙蝠)中出现强烈表明它们是自然起源的,可能是通过重组获得的。”
因此,这些数据有力地表明了SARS-CoV-2的自然起源。
此外,研究团队再次确定了RmYN02的蝙蝠宿主为马来菊头蝠(Rhinolophus malayanus),和一种马来菊头蝠标本中获得的序列(GenBank accession MK900703 )100%一致。
马来菊头蝠和中华菊头蝠都广泛分布于中国西南部和东南亚地区。论文中提到,一般来说,这些蝙蝠不会长距离迁移,群居性很强,很可能生活在同一个洞穴中,这可能会促进它们之间的病毒交换和重组的发生。
值得注意的是,科学家从肛门拭子中发现了RaTG13,从粪便中发现了RmYN02。因此,粪便是蝙蝠将病毒传播给其他动物,特别是能够利用洞穴环境的物种的一种简单但可行的方法。
责任编辑:宋璟
(原标题:细胞子刊:中国团队发现新蝙蝠冠状病毒,力证新冠起源于自然)